はじめに
エピゲノム入門の記事でもご紹介の通り、エピゲノムは、塩基配列の変化を伴わない後天的な化学的修飾により、遺伝子の発現を変化させる機構を指します。これらの修飾の代表的なものとしては、DNAメチル化、ヒストン修飾やクロマチン構造、non-cording RNAなどが挙げられます。本稿では、DNAメチル化と遺伝子制御に焦点を当て、エピジェネティックな修飾の理解を深めます。
DNAメチル化の概要
DNAメチル化は、DNAメチル基転移酵素により、シトシン塩基にメチル基が付加される化学的修飾で、主にCpGサイト(シトシンとグアニンが連続する配列)に起こります。DNAメチル化は、遺伝子のプロモーターやエンハンサー領域において、転写因子の結合を阻害し、遺伝子の発現を抑制する役割を果たします。一方で、遺伝子領域全体においては、DNAメチル化は遺伝子の安定性や構造を維持する役割も持っています。重要な点として、DNAメチル化は可逆性があり、メチル化過程と脱メチル化過程が存在するという点が挙げられます。
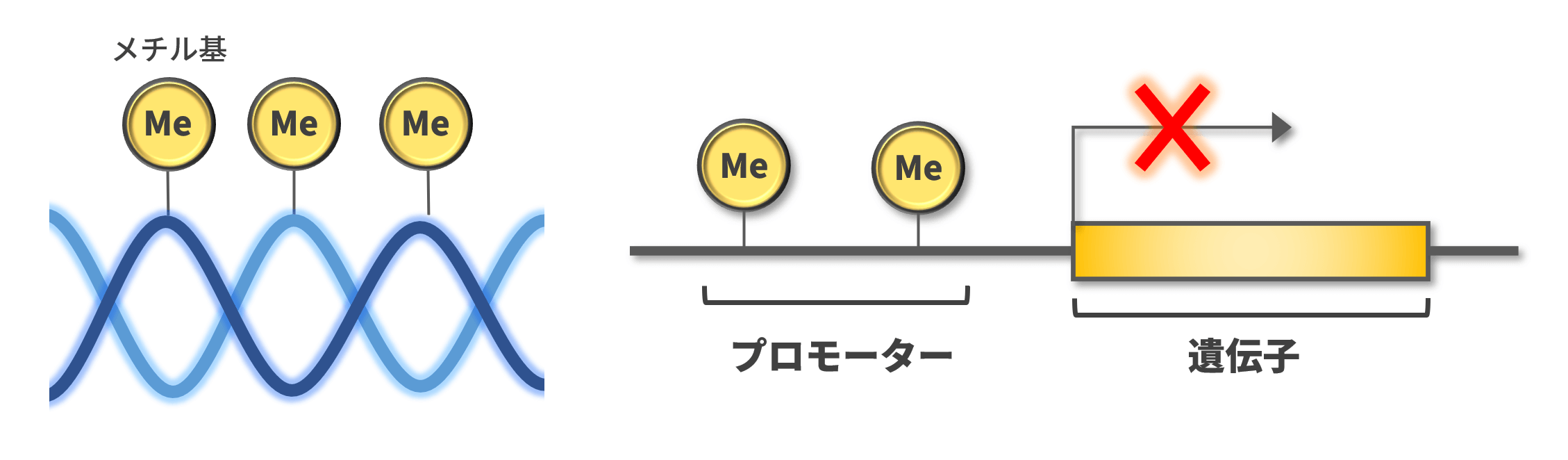
DNAメチル化と遺伝子制御
DNAメチル化は、遺伝子の発現制御において重要な役割を果たします。メチル化されたCpGは、転写因子の結合を阻害し、遺伝子の発現を抑制することが一般的です。また、メチル化されたDNAは、ヒストン脱アセチル化酵素(HDAC)などの転写抑制因子を引き寄せ、クロマチン構造を変化させ、転写をさらに抑制します。また、DNAメチル化には、維持メチル化機構が存在します。DNAメチル基転移酵素のひとつであるDNA methyltransferase 1(DNMT1)は、メチル化された親鎖の対になる新生DNA鎖にメチル基を付加することで、細胞分裂時にメチル化パターンを保持し継承します。
DNA脱メチル化
DNA脱メチル化は、DNAからメチル基が取り除かれる過程で、遺伝子の発現を促進することが一般的です。脱メチル化には、受動的な脱メチル化と能動的な脱メチル化があり、受動的な脱メチル化は、維持メチル化機構を抑制することで細胞分裂時にメチル化パターンの保持が出来なくなり、徐々に希釈されていくことで起こります。対して、能動的な脱メチル化は、主に、DNA脱メチル化酵素(TETファミリー)によって産生されます。TET酵素は、α-ケトグルタル酸(α-KG)を補酵素として5-メチルシトシン(5mC)を水酸化して5-ヒドロキシメチルシトシン(5hmC)に変換します。5hmCはさらに他の酸化産物へと変換され、最終的にこの酸化産物がDNAから取り除かれることで、非メチル化状態に戻ります。
バイアブルイエローアグーチマウスを用いた研究
バイアブルイエローアグーチマウスは、エピジェネティクスと遺伝子制御に関連する研究でよく使用されるモデル生物です。バイアブルイエローアグーチマウスは、Agouti遺伝子というマウスの毛色を制御するために働く遺伝子の上流に、プロモーター活性を持つレトロトランスポゾンが挿入されています。このレトロトランスポゾンがが高メチル化状態の場合、マウスは黒色または褐色の毛を持ちます。一方、この遺伝子が低メチル化状態の場合、マウスは黄色の毛を持つようになります。
2003年 Robert A. Waterlandらは、母マウスが妊娠中に摂取する栄養素が、子マウスのAgouti遺伝子のメチル化状態に影響を与えることを報告しました。例えば、葉酸やビタミンB12などのメチル化に関与する栄養素を含む食事を摂取すると、子マウスのAgouti遺伝子のメチル化が増加し、黒色または褐色の毛を持つマウスが生まれることが多くなります。逆に、これらの栄養素が不足していると、脱メチル化が起こり、黄色の毛を持つマウスが生まれることが多くなります。このアグーチマウスの実験結果は、環境要因がDNAメチル化を変化させ遺伝子発現に影響を与える可能性があること、さらにDNAメチル化が記憶され子孫に継承される可能性があることを示唆しており、今日まで興味深い研究のひとつとして挙げられています。

まとめ
エピジェネティックな修飾の一つであるDNAメチル化は、メチル化機構、メチル化維持機構、脱メチル化機構など、可逆性を伴い複雑に制御されつつ、遺伝子の発現制御に重要な役割を果たしています。
レリクサのサービスなら
丁寧なビフォアサポート・アフターサポート
15名以上の博士研究者が在籍。高い専門性と丁寧なサービスで、最後までご満足いただけるサービスを提供します。
リーズナブルで高品質なシーケンス
競争力のある複数の国内外シーケンスプロバイダーと提携し、低価格・高品質なデータ取得を実現しています。データ解析をご自身で実施するお客様からも選ばれています。
カスタム実験・オーダーメイド解析・統合解析にも対応
ご研究目的やご要望の図版、参考文献等に応じて、標準サービスメニュー外のカスタム実験・カスタム解析・マルチオミクス解析・統合解析も承ります。
参考文献
Jones, P. A. (2012). Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nature Reviews Genetics, 13(7), 484-492.
Deaton, A. M., & Bird, A. (2011). CpG islands and the regulation of transcription. Genes & Development, 25(10), 1010-1022.
Cedar, H., & Bergman, Y. (2009). Linking DNA methylation and histone modification: patterns and paradigms. Nature Reviews Genetics, 10(5), 295-304.
Bestor, T. H., Edwards, J. R., & Boulard, M. (2015). Notes on the role of dynamic DNA methylation in mammalian development. Proceedings of the National Academy of Sciences, 112(22), 6796-6799.
Wu, X., & Zhang, Y. (2017). TET-mediated active DNA demethylation: mechanism, function and beyond. Nature Reviews Genetics, 18(9), 517-534.
Egger, G., Liang, G., Aparicio, A., & Jones, P. A. (2004). Epigenetics in human disease and prospects for epigenetic therapy. Nature, 429(6990), 457-463.
株式会社Rhelixa(レリクサ)について
当社は最先端のゲノム・エピゲノム解析で培ってきた技術を活用して、生物学・医学・薬学領域における基礎研究や製品・ソリューションの開発、またはそれらの受託業務を行っています。次世代シーケンサーにより得られるエピゲノムデータの他、ゲノムやトランスクリプトーム、メタゲノムデータを組み合わせた統合的なデータ解析により、細胞制御の詳細なメカニズムの予測や精度の高いマーカーの探索を行います。また、研究開発のあらゆる場面で必要となるデータの統計解析や図版作成を基礎知識を必要とせず誰もが手元で実現できる環境を提供しています。